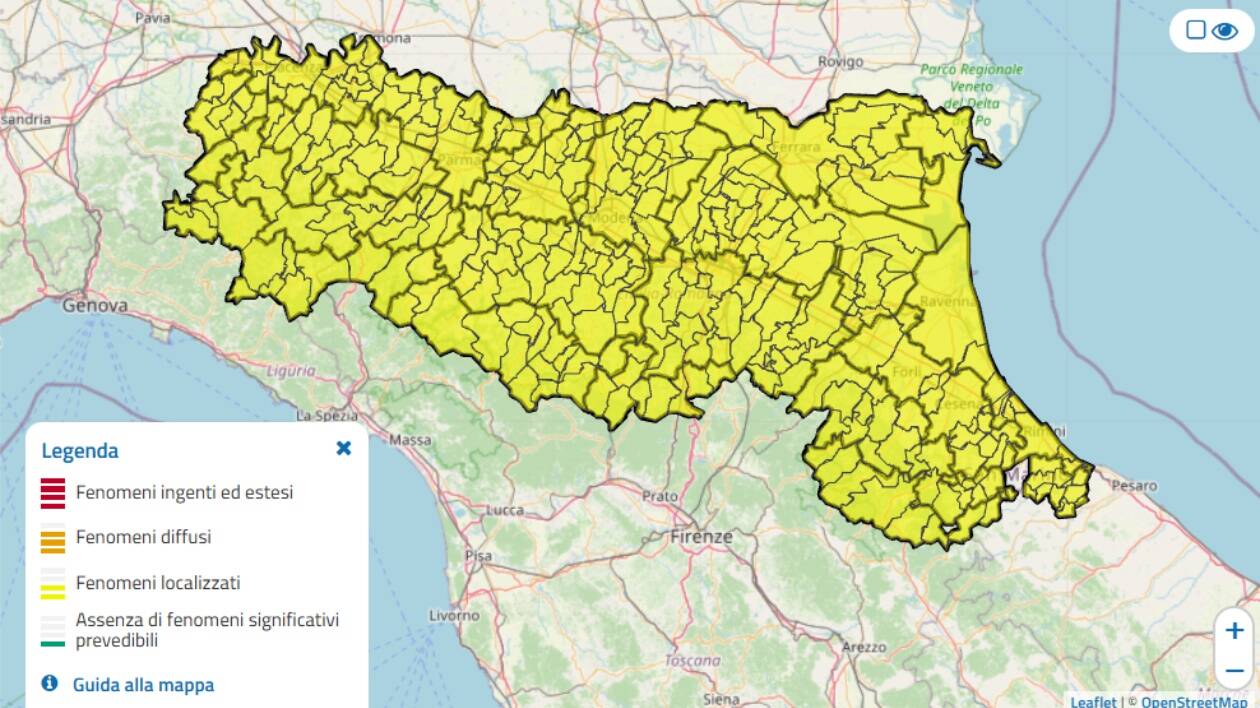
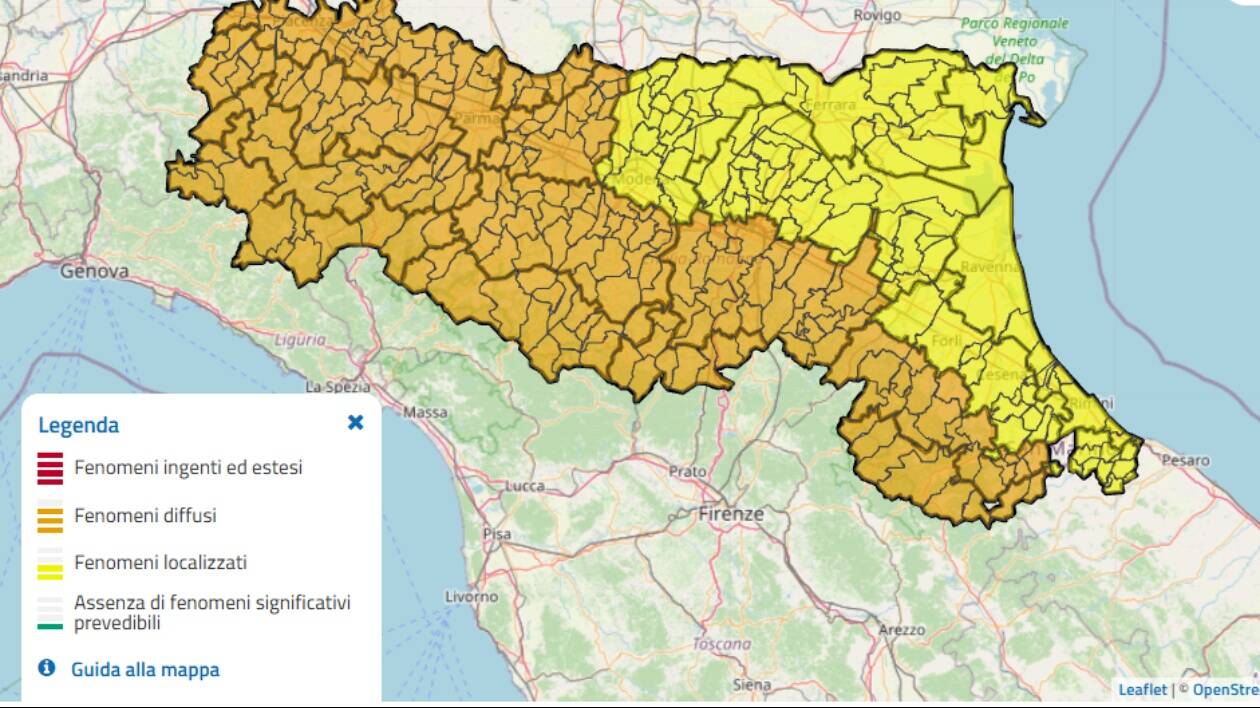
Dal 28 dicembre i tamponi di 23 pazienti, rientrati dal Regno Unito o da altri Paesi esteri a rischio, sono stati sottoposti al sequenziamento whole genome (intero genoma) del virus. Un’operazione particolarmente complessa, che richiede quattro giorni e mezzo di analisi, dalle quali sono emersi i 10 portatori della variante. Le condizioni dei pazienti sono buone, tutti sono stati posti in isolamento e sono stati tracciati i loro contatti. A spiegare come avviene l’analisi, è lo stesso professor Sambri: “In primo luogo prepariamo ‘manualmente’ l’RNA del virus, che poi viene letto da un’apposita apparecchiatura fino a 30.000-40.000 volte per stabilire la sequenza del genoma virale, un processo che richiede oltre 4 giorni. Al termine, attraverso software di analisi bionformatica, viene stabilita la sequenza vera e propria, e questo processo richiede un tempo ulteriore di alcune ore. L’enorme mole di dati è usata, sempre con software dedicati, per stabilire se la sequenza è relativa al virus ‘classico’, ad una delle varianti note, o eventualmente ad una nuova variante”. Un’operazione di estrema accuratezza, ma anche complessità, e quindi con tempi di esecuzione importanti. “Stiamo acquisendo- aggiunge Sambri- un’apparecchiatura robotica che ci consentirà di portare da 16 a 96 il numero di campioni analizzabili contemporaneamente, ma per ogni serie analitica i tempi resteranno comunque simili a quelli attuali”. Intanto, dopo la circolare ministeriale del 30 gennaio, la Regione ha deciso di ampliare le categorie di pazienti sui quali effettuare le sequenziazioni per cercare la presenza di varianti: saranno coinvolti anche coloro che fanno rientro da Portogallo, Brasile e Sudafrica, oltre a pazienti che presenteranno particolari criteri clinici.
Covid, in Emilia Romagna 10 contagiati dalla variante inglese

Il risultato è emerso dopo la?complessa analisi di sequenziazione?necessaria per l'individuazione di varianti virali
Dal 28 dicembre i tamponi di 23 pazienti, rientrati dal Regno Unito o da altri Paesi esteri a rischio, sono stati sottoposti al sequenziamento whole genome (intero genoma) del virus. Un’operazione particolarmente complessa, che richiede quattro giorni e mezzo di analisi, dalle quali sono emersi i 10 portatori della variante. Le condizioni dei pazienti sono buone, tutti sono stati posti in isolamento e sono stati tracciati i loro contatti. A spiegare come avviene l’analisi, è lo stesso professor Sambri: “In primo luogo prepariamo ‘manualmente’ l’RNA del virus, che poi viene letto da un’apposita apparecchiatura fino a 30.000-40.000 volte per stabilire la sequenza del genoma virale, un processo che richiede oltre 4 giorni. Al termine, attraverso software di analisi bionformatica, viene stabilita la sequenza vera e propria, e questo processo richiede un tempo ulteriore di alcune ore. L’enorme mole di dati è usata, sempre con software dedicati, per stabilire se la sequenza è relativa al virus ‘classico’, ad una delle varianti note, o eventualmente ad una nuova variante”. Un’operazione di estrema accuratezza, ma anche complessità, e quindi con tempi di esecuzione importanti. “Stiamo acquisendo- aggiunge Sambri- un’apparecchiatura robotica che ci consentirà di portare da 16 a 96 il numero di campioni analizzabili contemporaneamente, ma per ogni serie analitica i tempi resteranno comunque simili a quelli attuali”. Intanto, dopo la circolare ministeriale del 30 gennaio, la Regione ha deciso di ampliare le categorie di pazienti sui quali effettuare le sequenziazioni per cercare la presenza di varianti: saranno coinvolti anche coloro che fanno rientro da Portogallo, Brasile e Sudafrica, oltre a pazienti che presenteranno particolari criteri clinici.
Da anni Lapressa.it offre una informazione indipendente ai lettori, senza nessun finanziamento pubblico. La pubblicità copre parte dei costi, ma non basta. Per questo chiediamo a chi quotidianamente ci segue di concederci un contributo. Anche un piccolo sostegno, moltiplicato per le decine di migliaia di lettori, è fondamentale.

Escherichia coli in Romagna, non solo Cesenatico: non balneabili 17 tratti di mare: da Milano Marittima a Cattolica

Modena: dovevano rifornire di merce il supermercato, in realtà la rubavano
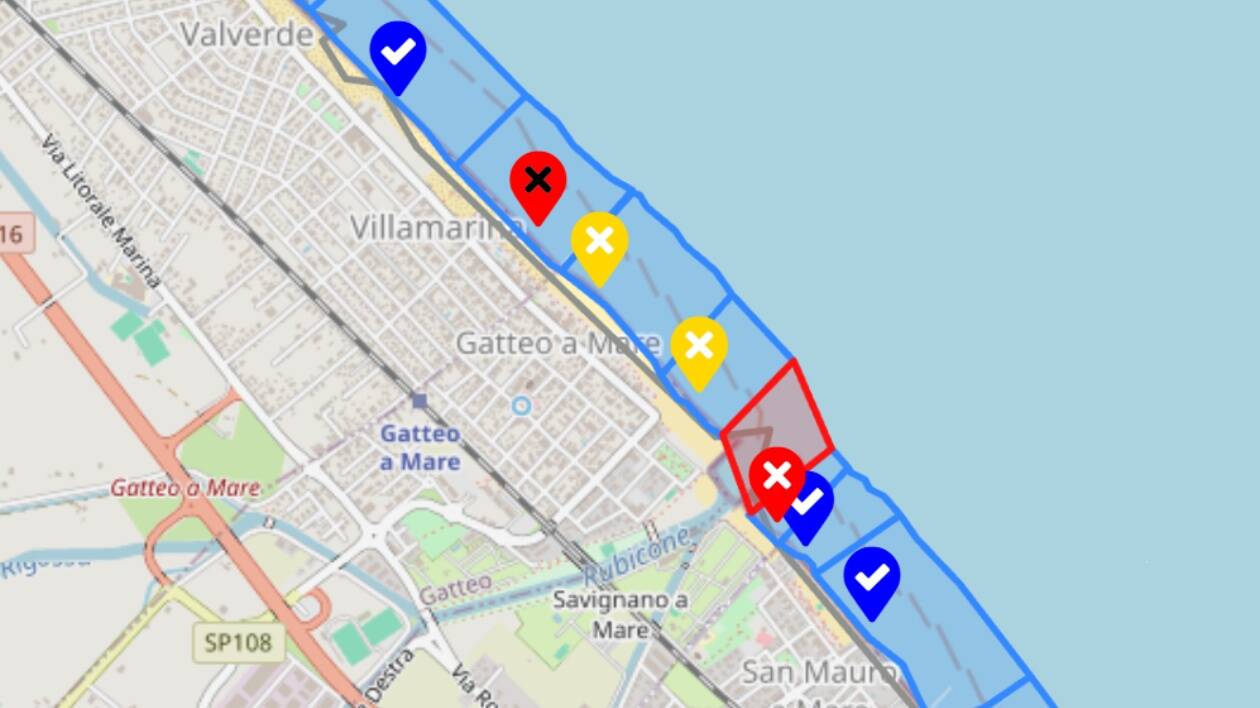
Romagna, escherichia coli superiore al valore limite: divieto di balneazione a Cesenatico

Escherichia coli ed Enterococchi: da Rimini a Riccione ecco i divieti di balneazione
Articoli Recenti
Ex Amcm, spaccio, degrado e bivacchi: qui è ancora terra di nessuno

Via Canaletto Sud chiude per la terza volta per lavori sulla filovia e per più di un mese

Rogo alla Dsv a Modena, l'azienda: 'Stiamo collaborando per chiarire le cause'

Modena, apertura della stagione di caccia: otto multe
 (1).jpg)